Análise Organóide
pelo Dr. Philip Seidel – Gerente de Marketing de Produto Software de Ciências da Vida ZEISS Microscopy – conteúdo Extraído do site ZEISS – empresa que representamos no estado do Rio de Janeiro
ABSTRATO
Da Imagem aos Resultados | Análise Organóide
Nesta nova série “Da imagem aos resultados”, explore vários estudos de caso que explicam como obter resultados de suas amostras exigentes e imagens adquiridas de maneira eficiente. Para cada estudo de caso, destacamos diferentes amostras, sistemas de imagem e questões de pesquisa.
Neste terceiro episódio, mostramos um experimento de imagem simples realizado em organoides intestinais tratados com e sem uma droga inibidora de Wnt, com o objetivo experimental de estudar o papel da sinalização de Wnt na formação de organoides .
Estudos chaves:
- Como estudar o papel da sinalização Wnt na formação de organoides
- Como usar o aprendizado de máquina para segmentar camadas externas de células organoides
- Como realizar a segmentação do núcleo e do corpo celular
Visão geral do estudo de caso
| Amostra | Organoides intestinais |
|---|---|
| Tarefa | Estude o papel da sinalização Wnt na formação de organoides |
| Resultados | Análise de um experimento organoide |
| Sistema | ZEISS Celldiscoverer 7 |
| Programas | arivis Vision4D® |

Introdução
A pesquisa em Biologia do Desenvolvimento visa entender os eventos e sinais de sinalização que ocorrem durante o desenvolvimento de organismos e órgãos vivos. Historicamente, a Biologia do Desenvolvimento dependia fortemente de organismos modelo complexos, como sapos com garras, moscas da fruta ou camundongos. Hoje, pesquisadores interessados no desenvolvimento de órgãos utilizam cada vez mais os organoides. Estes são sistemas de modelos tridimensionais artificiais que podem imitar a composição celular e a arquitetura dos tecidos dos órgãos, além de serem mais fáceis de manter e manipular experimentalmente.
Os organoides intestinais (intestinais) tornaram-se ferramentas indispensáveis para estudar tanto o desenvolvimento normal do intestino quanto os mecanismos que levam a morbidades (por exemplo, doença inflamatória intestinal). Os organoides intestinais são cultivados a partir de células-tronco intestinais únicas. Com os sinais de sinalização adequados aplicados, eles eventualmente formam organoides que consistem em uma única camada de enterócitos (células intestinais diferenciadas) em torno de um lúmen oco que se assemelha ao lúmen de um intestino real (Figura 1A).
A via Wnt é uma via de sinalização bem conhecida que regula o desenvolvimento e a manutenção do intestino. As funções e efeitos do Wnt são muito complexos e dependentes do contexto (para leitura detalhada, consulte a referência abaixo). Simplificando, Wnt contribui para a manutenção de células-tronco de tecidos saudáveis e a transição e diferenciação de células-tronco em enterócitos maduros (células de tecidos intestinais). Por outro lado, a atividade excessiva de Wnt (por exemplo, por mutações genéticas) contribui para o câncer intestinal.
Nesta história de aplicação, mostramos um experimento de imagem simples realizado em organoides intestinais tratados com e sem uma droga inibidora de Wnt, com o objetivo experimental de estudar o papel da sinalização de Wnt na formação de organoides.

- Figura 2A : Varredura geral de organoides (campo amplo).

- Figura 2B : Identificação das áreas de interesse.

- Figura 2C : Varredura confocal detalhada usando o detector Airyscan.
Material e métodos
Para este experimento, as células-tronco intestinais foram primeiro equipadas com proteínas fluorescentes Histone2B-RFP e Mem9-GFP para marcar núcleos e membranas celulares. Células-tronco intestinais isoladas foram deixadas crescer em organoides por 5 dias na presença ou ausência do inibidor da via de sinalização Wnt IWP-2. Os organoides foram então fixados e corados com anticorpo para Aldolase B, que é um marcador para enterócitos diferenciados, e contrastados com DAPI (para detecção de núcleo).
A aquisição de imagens foi realizada usando um ZEISS Celldiscoverer 7 confocal que combina os modos de imagem de campo amplo e confocal. Organoides únicos foram adquiridos com ampliação de 20X com pilhas de imagens abrangendo toda a profundidade do organoide.
Para a aquisição de muitos organoides individuais foi utilizado o módulo “Aquisição Guiada” da ZEISS ZEN (edição azul). Este é um fluxo de trabalho de imagem automatizado que consiste em três partes. Uma grande varredura de visão geral com baixa ampliação (Figura 2A). Um pipeline de análise de imagem para identificar áreas de interesse – neste caso, organoides individuais na imagem geral (Figura 2B) e uma varredura detalhada de todas as posições identificadas (Figura 2C).
A varredura geral foi realizada com uma ampliação de 2,5x no modo de campo amplo baseado em câmera. Para varreduras detalhadas (ampliação de 20x), as pilhas de imagens abrangendo a profundidade organoide completa foram capturadas no modo confocal usando o detector Airyscan.

Figura 2D : Imagens gerais de organoides que foram tratados com e sem inibidor de Wnt. As imagens mostram que a inibição de Wnt altera a morfologia dos organoides, incluindo tamanho e forma. Os organoides tratados com controle são maiores e têm uma forma irregular.


Figura 2E : Os organoides tratados com controle são maiores e têm uma forma irregular. A Figura 2E mostra como a inibição de Wnt provavelmente reduz a expressão de Aldolase B.


arivis Vision4D
arivis Vision4D® é um software modular para trabalhar com imagens multicanal 2D, 3D e 4D de tamanho quase ilimitado, altamente escalável e independente dos recursos do sistema local. Muitos sistemas modernos de microscópio, como confocal de alta velocidade, folha de luz / SPIM, super-resolução, microscopia eletrônica ou instrumentos de raios-X, podem produzir grandes quantidades de dados de imagem. arivis Vision4D lida com esses conjuntos de dados sem restrições e em tempo relativamente curto.

Processamento de software
Primeiro, os dados brutos da imagem de aquisição (um arquivo .czi por organoide) foram importados em lote para um único arquivo Vision4D. Para amostras de controle simulado e inibidor de Wnt, 30 organoides foram incluídos na análise. Para reduzir o tamanho dos dados, as imagens foram transformadas em 8 bits e agrupadas em 2×2 em dimensões xy.
O conjunto de dados de imagem bruta foi então processado por meio de normalização de imagem nos canais H2B-RFP, Mem9-GFP e DAPI para explicar as diferenças de intensidade entre organoides e as diferenças de intensidade do sinal devido à profundidade da imagem.
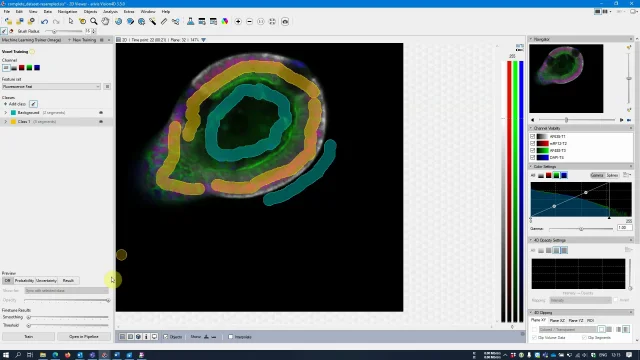
Em seguida, a segmentação de aprendizado de máquina foi realizada para segmentar a camada externa de células organoides. O lúmen organoide foi em seguida determinado preenchendo inclusões na segmentação da camada de células organoides.
Os núcleos foram segmentados com a função blob finder dos canais H2B-RFP e DAPI. Os núcleos dentro da camada de células organoides e o lúmen organoide foram separados em dois grupos de objetos com base nas distâncias do objeto ao lúmen organoide. Os corpos celulares foram segmentados através da região que cresce a partir de objetos nucleares dentro da camada de células organoides.
Finalmente, para facilitar uma melhor análise estatística, todos os grupos de objetos foram estratificados para organoides únicos. O pipeline está disponível para download e teste no final desta história do aplicativo.
Execução em arivis Vision4D
7:12
Aprendizado de máquina
Neste vídeo tutorial, aprenda como usar o aprendizado de máquina no arivis Vision4D para analisar organoides, que foram fotografados no microscópio confocal ZEISS Celldiscoverer 7.
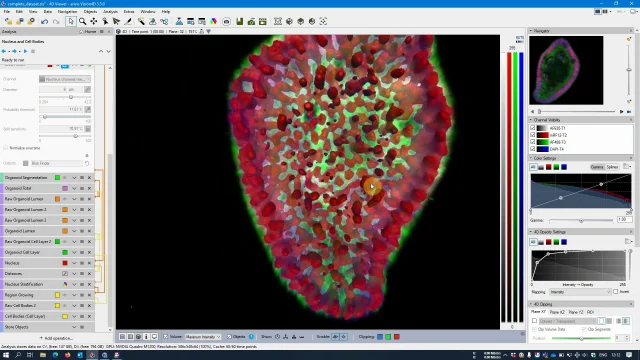
7:39
Segmentação do núcleo e do corpo celular
Neste vídeo tutorial, continue com a análise de organoides usando arivis Vision4D. No tutorial anterior, usamos aprendizado de máquina para medir o volume total de um organoide. Agora aprenda a segmentar os núcleos individuais usando a operação Blob Finder e modele os limites da célula usando a operação Region Growing.
Validação
Em seguida, verificamos a validade e a qualidade das diferentes segmentações aplicadas durante a análise. A camada de células organoides e o lúmen organoide foram segmentados com o segmentador de aprendizado de máquina. Empregar o aprendizado de máquina leva a resultados de segmentação superiores em comparação com a segmentação convencional baseada em limites. Permitiu a discriminação entre células da camada celular (incluídas nos objetos) e lúmen (excluídas dos objetos) com base na textura complexa da imagem (Figura 3A).
Os núcleos celulares foram segmentados com segmentação localizador de blob. Isso permitiu a separação de núcleos de alta qualidade, apesar de estarem densamente empacotados em 3D e apesar das variações de intensidade. Ao estabelecer relações entre a camada de células organoides e o objeto lúmen, os núcleos foram então separados em núcleos da camada celular e núcleos luminais (Figura 3B). Os corpos celulares foram segmentados por região de crescimento dos núcleos da camada celular. Por filtragem de objetos, eles são bem restritos à camada de células organoides (Figura 3C).

Figura 3A : Camada de células organoides e segmentação do lúmen. Sobreposição de camada de células mostrada em verde, sobreposição de lúmen em amarelo.

Figura 3B : Núcleos na camada de células organoides e lúmen. Núcleos da camada celular mostrados em vermelho, núcleos luminais mostrados em amarelo.

Figura 3C : Corpos celulares na camada de células organoides. Núcleos da camada celular mostrados em vermelho, corpos celulares da camada celular mostrados em verde.
Resultados
Tamanho e arredondamento dos organoides
Tendo segmentado e validado todos os objetos relevantes de interesse, agora podemos pular para a análise. Por causa da variação natural dentro dos esferoides e para permitir uma análise estatística simples, todas as análises são preparadas como gráficos de bigodes de caixa (representando pontos de dados únicos e suas médias e desvios padrão), comparando organoides de ambos os grupos experimentais, onde cada ponto é o dados de um organoide.
Primeiro, os volumes do organoide completo (Figura 4A), a camada externa de células organoides (Figura 4B) e o lúmen organoide (Figura 4C) foram analisados. Há uma tendência para volumes maiores e principalmente uma maior dispersão de volumes no grupo controle, sugerindo que a inibição de Wnt interfere no crescimento adequado dos esferóides. No entanto, nenhuma dessas tendências foi significativa em um teste t estatístico. Tempos de incubação mais longos ou incluir mais organoides na análise provavelmente ajudariam a obter resultados mais conclusivos.
Também observamos na imagem de visão geral inicial (Figura 2A) que os organoides tratados com controle formaram mais formas amorfas, enquanto os organoides tratados com o inibidor de Wnt permaneceram esféricos. O Vision4D oferece vários parâmetros morfológicos para analisar tais observações. A Figura 4D mostra uma queda significativa de “arredondamento” em amostras tratadas com controle, assim, a inibição de Wnt de fato interfere na formação de formas organoides amorfos.

Figura 4A : O volume dos organoides completos. Pontos de dados únicos, média e desvio padrão são descritos. O valor p do teste t estatístico é mostrado.

Figura 4B : O volume das camadas de células organoides. Pontos de dados únicos, média e desvio padrão são descritos. O valor p do teste t estatístico é mostrado.

Figura 4C : O volume do lúmen interno organoide. Pontos de dados únicos, média e desvio padrão são descritos. O valor p do teste t estatístico é mostrado.

Figura 4D : A redondeza de organoides completos. Pontos de dados únicos, média e padrão são o desvio representado. O valor p do teste t estatístico é mostrado.
Números de células em diferentes compartimentos organoides
Em seguida, como outro marcador de crescimento organoide, avaliamos o número de células nos diferentes compartimentos organoides com base na contagem de objetos do núcleo. A quantificação foi realizada para todos os núcleos, com os núcleos da camada celular e os núcleos luminais processados independentemente. Essas duas classes têm destinos completamente diferentes dentro do organoide em crescimento. As células luminais atuam apenas como um suporte transitório e eventualmente morrem por apoptose, enquanto as células da camada externa formam o tecido epitelial funcional do organoide.
Os números de células são mostrados para o organoide total (Figura 5A), a camada externa de células organoides (Figura 5B) e o lúmen organoide (Figura 5C). Para todos os três grupos, há um aumento significativo no número de células para organoides tratados com controle em comparação com organoides expostos à inibição de Wnt (p <0,05 cada em testes t estatísticos). Isso indica novamente que a inibição de Wnt interfere com o crescimento organoide adequado. Quanto aos volumes organoides, a dispersão dos valores (o desvio padrão) é considerável. Portanto, a análise provavelmente se beneficiaria da análise de tamanhos de amostra maiores (mais esferóides na análise).

Figura 5A : Os números de células dos organoides completos. Pontos de dados únicos, média e desvio padrão são descritos. O valor p do teste t estatístico é mostrado.

Figura 5B : Os números de células das camadas de células organoides. Pontos de dados únicos, média e padrão são o desvio representado. O valor p do teste t estatístico é mostrado.

Figura 5C : Os números de células do lúmen interno organoide. Pontos de dados únicos, média e padrão são o desvio representado. O valor p do teste t estatístico é mostrado.
Expressão de Aldolase B como Marcador para Diferenciação de Enterócitos
Tendo descrito as características organoides morfológicas básicas nas seções anteriores, agora nos voltamos para a análise da expressão da Aldolase B. A aldolase B é um marcador de diferenciação de enterócitos. Durante a maturação organoide, as células-tronco intestinais devem se transformar progressivamente em células diferenciadas. Como resultado, o aumento da expressão da Aldolase B é um indicador para a maturação organoide bem-sucedida.
Dois de nossos grupos de objetos poderiam teoricamente ser adequados para a análise: (1) os núcleos da camada celular e (2) os corpos celulares da camada celular. Conforme mostrado na Figura 6A, a Aldolase B localiza-se principalmente no citosol, tornando os objetos do corpo celular os mais adequados para análise. O Vision4D permite extrair intensidades de canais de diferentes camadas hierárquicas. Aqui, mostramos aqui a soma da expressão de Aldolase B para o organoide completo (Figura 6B) e as intensidades médias de Aldolase B de uma única célula medidas independentemente em cada célula (Figura 6C). Em ambos os casos, há um aumento forte e significativo (p<0,001 em testes T estatísticos) em organoides que foram tratados de forma simulada em comparação com organoides tratados com inibidor de Wnt. Isso adiciona mais evidências de que a inibição de Wnt interfere na maturação organoide.

Figura 6A : Localização da expressão da Aldolase B nos organoides. A expressão da aldolase B (cinza) está localizada nos corpos celulares inteiros (verde) e não nos núcleos (vermelho).

Figura 6B : Expressão organoide Aldolase B total. Pontos de dados únicos, média e desvio padrão são descritos. O valor p do teste t estatístico é mostrado.

Figura 6C : Intensidade média celular média da Aldolase B. Pontos de dados únicos, média e padrão são o desvio representado. O valor p do teste t estatístico é mostrado.
Determinando células positivas para aldolase B como uma leitura alternativa
Na última seção, nos concentramos em medir um espectro contínuo de expressão da Aldolase B; no entanto, isso tem uma séria desvantagem. Ele não considera que, mais realisticamente, as células sejam “positivas” ou “negativas” para Aldolase B, como pode ser observado em uma seção transversal típica de organoides (Figura 7A). Portanto, uma estratégia de análise mais adequada estratifica as células em grupos positivos e negativos para Aldolase B e, em seguida, avalia a fração de células positivas dentro de um organoide.
Usando esta abordagem, deve ser definido um limiar que separe as populações positivas e negativas. A Figura 7B mostra a distribuição bimodal da intensidade da Aldolase B em todas as células no conjunto de dados, com máximos em intensidades médias de pixel de 10 e 30, respectivamente. Selecionamos uma intensidade média de pixel de 15 como limiar para células positivas para Aldolase B. A aplicação desse limite gera células positivas e negativas que combinam bem com a impressão visual da distribuição de Aldolase B na seção transversal de exemplo (Figura 7A). Os resultados são mostrados como o total de células positivas por organoide (Figura 7C) e como a porcentagem de células positivas por organoide (Figura 7D). Mais uma vez, os organoides tratados com controle tinham significativamente mais células positivas para Aldolase B, indicando melhor maturação do organoide.

Figura 7A : Localização da expressão da Aldolase B nos organoides. A expressão da aldolase B (cinza) está localizada nos corpos celulares inteiros (verde) e não nos núcleos (vermelho).

Figura 7B : Distribuição média da intensidade da Aldolase B de células únicas. Observe a curva de distribuição bimodal e o limiar definido entre os dois máximos.

Figura 7C : Número de células positivas para Aldolase B por organoide. Pontos de dados únicos, média e desvio padrão são descritos. O valor p do teste t estatístico é mostrado.

Figura 7D : Porcentagem de células positivas para Aldolase B por organoide. Pontos de dados únicos, média e desvio padrão são descritos. O valor p do teste t estatístico é mostrado.
Nota Adicional – Análise de
Depois que um pipeline foi criado e otimizado no arivis Vision4D por meio de testes em um conjunto de imagens de amostra, é possível ampliar a análise usando a plataforma VisionHub baseada em servidor. Comoarivis VisionHubé acessado pela web, os usuários podem facilmente fazer upload de pipelines para seus conjuntos de dados, executar análises em lote e, em seguida, ter os resultados prontamente disponíveis na interface de usuário baseada na web.
Neste estudo, 60 amostras de organoides foram analisadas, mas seria possível analisar muito mais. o arivis VisionHub observa imagens recém-adquiridas sendo exportadas do microscópio. Depois que o conjunto de dados é totalmente exportado, a miniatura se torna visível na interface do usuário (Figura 8A). Os usuários podem fazer upload de seus pipelines e simplesmente selecionar conjuntos de dados para análise (Figura 8B). Quando a análise estiver concluída, os resultados estarão disponíveis como marcações de objetos no visualizador (Figura 8C) e as tabelas de dados também estarão disponíveis para download.

Figura 8A : Conjuntos de dados no VisionHub. Os usuários podem selecionar conjuntos de dados para análise de alto rendimento.

Figura 8B : Agendador de análise. Os trabalhos de análise são enfileirados.

Figura 8C : Resultados no VisionHub. Todos os resultados da análise estão disponíveis no VisionHub.
Resumo
Nesta história de aplicação, apresentamos uma abordagem para um estudo organoide empregando um ZEISS Celldiscoverer 7 e arivis Vision4D para análise de imagem.
Os organoides exigem muito das técnicas analíticas por causa de sua arquitetura tecidual bastante complexa (conjuntos de dados 3D que consistem em uma camada externa de células e um lúmen interno) e pela multiplicidade de marcadores de diferenciação que podem ser aplicados (volume e forma organoides, número de células e -análise da expressão celular do tecido). Destacamos como tudo isso pode ser feito com a funcionalidade integrada do Vision4D.
Neste experimento, encontramos vários resultados que podem destacar o papel da sinalização Wnt na organogênese intestinal. Pudemos validar que organoides com sinalização Wnt inibida teriam volumes organoides diminuídos, uma forma esférica mais imatura, menos número de células e menos expressão de Aldolase B. A aldolase B como marcador de diferenciação de enterócitos foi reduzida tanto em termos de níveis de expressão total quanto em termos de frações celulares positivas para Aldolase B.
Deve-se notar que apenas 30 organoides por amostra foram analisados com o objetivo de demonstrar a análise de imagem com nosso software. Isso certamente não atende aos altos padrões que seriam necessários para um estudo profissional e conclusões estatisticamente relevantes. Ainda acreditamos que ter esse tipo de caso de uso do “mundo real” ajuda você a aprender sobre estratégias de análise de imagem que podem ser aplicadas aos seus próprios dados. Como sempre, o conjunto de dados e o pipeline de análise são fornecidos aqui para dar a você a oportunidade de testar o Vision4D por conta própria.
Como começar
Neste vídeo, aprenda como começar com o conjunto de dados deste estudo de caso e sua versão de avaliação do arivis Vision4D (ambos os downloads são fornecidos no site da zeiss.com). Veja como carregar uma imagem, carregar um pipeline e como iniciar sua análise.

Clique para download dos dados do estudo de caso